КОМПЬЮТЕРНОЕ МОДЕЛИРОВАНИЕ СТРУКТУРЫ АМИЛОЛИТИЧЕСКИХ ФЕРМЕНТОВ
Ковалева
Т.А., Кожокина О.М., Битюцкая Л.А., Дронов Р.В., Мельников Л.Ю.
(Воронеж)
Используя данные рентгеноструктурного анализа и
программу MolScript, методом компьютерного моделирования построена трехмерная
модель макромолекулы глюкоамилазы из Aspergillus awamori, что позволило
по-новому оценить многочисленные экспериментальные данные, полученные классическими
методами (электронная спектроскопия, ЯМР, ИК спектроскопия и т.д.). Показаны
особенности строения активного центра
данного фермента, обозначены функционально важные группы. Внесены дополнения в представления о
механизме катализа амилолитическими ферментами.
COMPUTER MODELING
OF STRUCTURE OF AMYLOLITIC FERMENTS
T.A.Kovaleva,
O.M.Kozhokina, L.A.Bitutskaya, R.V.Dronov, L.Y.Melnikov
(Voronezh)
Using XRD data and MolScript program by the means of
computer modeling 3d model of the Glucoamylase from Aspergillus awamori
macromolecule was produced. It allowed us to reevaluate numerous experimental
data which were produced by classical means (STM, NMR, IR-spectroscopy, etc.).
The properties of structure of affinity labeling of this ferment were shown,
also functional redundant groups were marked. There were made some complements
to the representations of catalysis by amylolitic ferment.
При изучении механизмов ферментативного катализа классическими методами (ИК и УФ- спектроскопия, электронная спектроскопия, ЯМР и т.д.) дополнительную информацию вносит компьютерное моделирование белковых молекул с использованием данных рентгеноструктурного анализа (РСА). Способность глобулярных белков образовывать качественные монокристаллы, в которых все атомы идентичны и ориентированы одинаковым образом, позволила изучать их с помощью метода кристаллографии. Известно, что данные рентгеноструктурного анализа всегда правильно отражают укладку белковой цепи, а в подавляющем большинстве случаев буквально воспроизводят активную конформацию фермента. Следовательно, они являются надежной и пока что единственной основой для количественного описания механизма каталитического акта на атомно-молекулярном уровне. Метод кристаллографии позволяет расшифровывать трехмерные структуры комплексов ферментов с ингибиторами, которые в той или иной мере соответствуют химическим стадиям катализа. Располагая рентгеноструктурными данными, одна часть которых отвечает реальному исходному состоянию фермента, а другая - фермент-ингибиторным комплексам, становится возможным воссоздание механизма каталитического акта, а, следовательно, можно проследить динамику поведения белковой макромолекулы [1]. Разрабатываемые в настоящее время программы компьютерного моделирования белковых молекул на основе результатов кристаллографии позволяют получать наглядные трехмерные изображения исследуемых объектов, и по-новому осмыслить данные экспериментальных исследований кинетико-термодинамических свойств ряда ферментов, полученные за последнее десятилетие, а, следовательно, выявить дополнительные аспекты молекулярных механизмов ферментативного превращения субстрата. В этой связи целью данной работы является анализ топологии амилолитических ферментов с использованием программы MolScript по данных рентгеноструктурного анализа. Повышенный интерес к амилазам обусловлен их широким применением в медицине, пищевой и легкой промышленности в качестве эффективных биокатализаторов [2,3]. Исследования физико-химических свойств, кинетико-термодинамических параметров, особенностей фермент-субстратных взаимодействий при осуществлении акта катализа амилолитическими ферментами способствовали существенному изменению и усовершенствованию многих существующих технологий и созданию новых. Однако, очевидным является недостаток информации о пространственной организации макромолекул ферментов данной группы.
Объектом наших экспериментальных исследований является глюкоамилаза из Aspergillus awamori, препарат Г-20Х производства Ладыжинского завода ферментных препаратов. Для определения активности глюкоамилазы использовали глюкозооксидазный метод. Определение общего количества белка в ферментном препарате проводили по методу Лоури [4]. Исследование соотношения типов вторичной структуры осуществляли методом инфракрасной спектроскопии на спектрометре Specord M-80 (Германия). Определение молекулярной массы глюкоамилазы, а также изучение четвертичной структуры фермента с использованием додецилсульфата натрия в концентрации 3,5×10-5 моль/л для разрушения надмолекулярного уровня организации, проводили с помощью гель-хроматографии на сефадексе G-200. Количественное определение SH-групп осуществляли методом, основанном на измерении прироста оптической плотности в области 255 нм при присоединении n-меркурибензоата к SH-группам. Для количественного определения S-S групп использовали метод, основанный на измерении оптической плотности (l=412нм.) при образовании нитротиобензоата. Определение числа ионогенных групп проводили на автоматической установке, разработанной на кафедре биофизики и биотехнологии ВГУ, путем сравнения кривых титрования белка. Изучение пространственной структуры глюкоамилазы осуществляли с использованием программы MolScript [5].
Программа MolScript используется для моделирования протеиновых и протеидовых структур. Исходными данными для программы является *.in файл, в котором указывается файл рентгеноструктурного анализа, объекты для вывода и точное их описание с помощью графических параметров. Для создания в первом приближении входных файлов MolScript используется программа MolAuto. Эта программа распознает вторичную структуру в файле pdb и выводит соответствующие команды для схематического описания данной структуры. MolAuto имеет несколько ключей, позволяющих влиять на свойства объектов. Программа не способна выбирать удобный ракурс, поэтому пользователю необходимо выбирать его самостоятельно. MolAuto может быть частью потока UNIX с программой MolScript: например для быстрого получения изображения структуры в режиме OpenGL. Каждый формат, поддерживаемый MolScript, обладает своими специфическими свойствами. Поэтому выбор формата производится с учетом установленного программного обеспечения и желаемого набора свойств полученной модели.
Программа MolScript использует одну фиксированную правостороннюю систему координат с ценой деления 1 ангстрем. Точка обзора зафиксирована и измениться не может. Для просмотра молекулы с разных ракурсов атомные координаты необходимо трансформировать, т.е. повернуть и/или транслировать в пределах системы координат. Молекулярные координаты считываются из pdb файла, а сами координаты атомов и остатков, входящих в данный файл, используются графическими командами. Координаты выбранных атомов трансформируются матрицей, определенной одной или несколькими операциями.
При изучении топологии для двумерных статических и трехмерных моделей проводилось распознавание субъединиц ферментов в разных масштабах по различным направлениям. Топологические аспекты рассматривались как для фрагментов, так и для целостных молекул амилолитических ферментов, что позволило ввести обозначения функционально значимых групп.
Классические методы изучения особенностей протекания ферментативного катализа позволяют рассматривать широкий круг вопросов. Так, экспериментальным путем нами было исследовано влияние различных физико-химических факторов на конформацию макромолекулы глюкоамилазы из Aspergillus awamori и связанных с ней механизмов активации и инактивации данного фермента; изучены кинетико-термодинамические аспекты реакции гидролиза крахмала [6,7,8]. Методом гель-хроматографии определена молекулярная масса глюкоамилазы, составляющая 106 кДа. Исследование аминокислотного состава глюкоамилазы показало, что молекула фермента содержит 26,8% аминокислотных остатков с алкильными боковыми цепями (Ala, Val, Leu, Met, Ile), определяющими высокую устойчивость фермента к органическим растворителям. Обнаружено высокое содержание в молекуле глюкоамилазы Ser и Thr. Результаты наших экспериментов и анализ данных литературы по секвенированию глюкоамилазы (А.Е. Алешин и др.,1994) позволяют сделать заключение о том, что молекула фермента имеет 6 SH-групп и 4 дисульфидных связи, стабилизирующих его третичную структуру. Изучение надмолекулярного уровня организации глюкоамилазы показало наличие четвертичной структуры, представленной двумя идентичными субъединицами с молекулярной массой 53600Да, обладающими каталитической активностью. Ее стабильность определяется водородными связями и гидрофобными взаимодействиями и может зависеть от небольших изменений в пространственной структуре каждой из взаимодействующих субъединиц. Причем четвертичная структура, по-видимому, обладает способностью передачи структурных перестроек от одной субъединицы к другой и является одним из факторов регуляции функциональной активности молекулы белка [9].
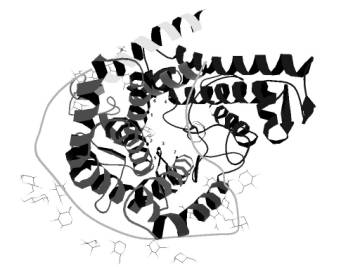
Рис. 1. Пространственная структура глюкоамилазы
Обозначения: 1 - Leu-58;2 - Leu-130; 3 - Leu-177; 4 – Leu-319; 5 – Trp-178; 6 – Trp-417; 7- Phe-187
Результаты исследований влияния n-меркурибензоата на каталитическую активность глюкоамилазы позволяют сделать заключение о том, что в состав активного центра данного фермента не входят имидазольная группа гистидина и SH-группы, а в гидролизе крахмала принимают участие карбоксильные группы аспарагиновой и глутаминовой кислот.
Используя данные рентгеноструктурного анализа [10] и программу MolScript, методом молекулярного моделирования показано, что активный центр глюкоамилазы расположен в сквозной полости (~ 1,5нм), ограниченной следующими аминокислотными остатками: Leu-58, Leu-130, Leu-177, Leu-319, Trp-178, Trp-417, Phe-187 (рис1).
Установлено, что в гидролизе крахмала принимают участие карбоксильные группы Asp-55, Glu-179, Glu-400 (рис.2).
Активный центр фермента располагается в щели, прикрытой двумя
кластерами молекул H2O: первый - в области Leu-58 (12 молекул H2O), второй – в углублении
активного центра (7 молекул). Показано, что с одной стороны щели активного
центра сосредоточены Asp-55
и Glu-179, а с
противоположной – Glu-400.
Результаты рентгеноструктурного
анализа (Алешин А.Е. и др, 1994 [10]) показали, что поверхность молекулы
глюкоамилазы гликозилирована: 10 аминокислотных остатков серина и треонина
имеют маннозу (Ser-453, Ser-455, Ser-459,
Thr-457 и др.).
Данный фермент характеризуется наличием N-терминального домена, состоящего из 440 аминокислотных остатков, O-гликозилированного участка, имеющего 70 аминокислот и C-терминального крахмалсвязывающего домена (100 аминокислот). Каталитический домен имеет 2 N-гликозилированных участка, причем контакт между N-гликозилированными цепями и полипептидом стабилизируется остатком маннозы с помощью водородной связи или ионизированными молекулами воды, чем и определяется стабильность фермента и возможность образования надмолекулярных структур. O-гликозилированный домен имеет остатки Gly перед и после C-конца, которые представляют собой изгибы, определяющие взаимодействие крахмалсвязывающего домена с каталитическим и соответствующую ориентацию молекулы субстрата. Гликозилирование предотвращает скопление молекул субстрата в связывающих центрах и обеспечивает стехиометрическое связывание.
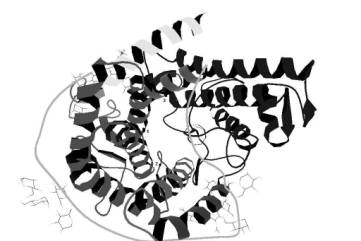
Рис. 2. Пространственная структура макромолекулы глюкоамилазы
Обозначения: 1 – Asp-55; 2 – Glu-179; 3 – Glu-400
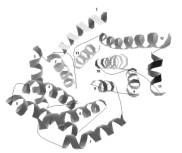
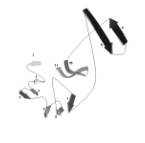
Методом компьютерного моделирования установлено, что для молекул глюкоамилазы характерна плотная упаковка гидрофобного ядра в виде 13 α-спиральных участков (рис.3), а так же антипараллельных β-структур, образующих 11 петель (рис.4).
|
Рис. 3. Топология α-спиралей в макромолекуле глюкоамилазы |
Рис. 4. Топология β-слоев в макромолекуле глюкоамилазы |
Во вторичной структуре присутствуют, кроме того, неупорядоченные участки в количестве 19 (рис.5). В процентном соотношении данных структур по протяженности, аморфные участки преобладают над остальными, что подтверждено также методом ИК-спектроскопии (таблица).
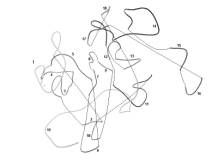
Рис. 5. Топология неупорядоченных участков в макромолекуле глюкоамилазы
Таблица. Протяженность участков
вторичной структуры в макромолекуле глюкоамилазы
|
a-спирали |
b-слои |
Неупорядоченные участки |
|
1: c 1 по 20 ост. 20 2: c 54 по 68 ост. 15 3: c 72 по 89 ост. 18 4: c 126 по 144 ост. 19 5: c 148 по 168 ост. 21 6: c 186 по 205 ост. 20 7: c 211 по 227 ост. 17 8: c 245 по 254 ост. 10 9: c 272 по 285 ост. 14 10: c 318 по 338 ост. 21 11: c 345 по 354 ост. 10 12: c 368 по 391 ост. 24 13: c 416 по 429 ост. 14 |
1: c 21 по 23 ост. 3 2: c 49 по 53 ост. 5 3: c 107 по 109 ост. 3 4: c 114 по 116 ост. 3 5: c 173 по 175 ост. 3 6: c 181 по 183 ост. 3 7: c 235 по 237 ост. 3 8: c 339 по 344 ост. 6 9: c 360 по 363 ост. 4 10: c 398 по 403 ост. 6 11: c 407 по 415 ост. 9 |
1: c 23 по 49 ост. 27 2: c 68 по 72 ост. 5 3: c 89 по 107 ост. 19 4: c 109 по 114 ост. 6 5: c 116 по 126 ост. 11 6: c 144 по 148 ост. 5 7: c 168 по 173 ост. 6 8: c 175 по 181 ост. 7 9: c 183 по 186 ост. 4 10: c 205 по 211 ост. 7 11: c 227 по 235 ост. 9 12: c 237 по 245 ост. 9 13: c 254 по 272 ост. 19 14: c 285 по 318 ост. 34 15: c 354 по 360 ост. 7 16: c 363 по 368 ост. 6 17: c 391 по 398 ост. 8 18: c 403 по 407 ост. 5 19: c 429 по 471 ост. 42 |
|
Всего 223 (44%) |
Всего 48 (9.5%) |
Всего 236 (46.5%) |
Таким образом, компьютерное моделирование позволяет получать дополнительную информацию о структуре белковых макромолекул и проводить расшифровку механизма ферментативного катализа.
В настоящее время продолжаются работы по проблемам изучения топологии различных амилолитических ферментов, таких как α-амилаза, β-амилаза, инулаза и др. Обсуждаются вопросы топологической самоорганизации исследуемых белковых макромолекул, определяемой взаимосвязью первичной и вторичной структуры.
Список литературы:
1.Попов Е.М. Структурно-функциональная организация белков. – М.: Наука, 1992.–358 с.
2.Галич И.П. Амилазы микроорганизмов. – Киев: Наук. думка, 1987. – 192 с.
3.Жеребцов Н.А. Амилолитические ферменты в пищевой промышленности. – М.:Лег. и пищ. пром-сть, 1984. – 160 с.
4.Кочетов Г.А. Практическое руководство по энзимологии. – М.:Высш. шк., 1980. – 272 с.
5.Kraulis P.
Avatar software AB 1998. http://www.avatar.se
6.Ковалева Т.А., Селеменев В.Ф., Гречкина В.Р. Некоторые свойства глюкоамилазы // Труды III Всесоюзной межуниверситетской конференции по физико-химической биологии. – Тбилиси, 1982. – С. 240-241.
7.Изучение некоторых свойств глюкоамилазы сорбционными и спектральными методами / Ковалева Т.А., Селеменев В.Ф., Плохих А.М. и др.// Теория и практика сорбционных процессов. – Воронеж, 1985. – Вып. 17.-С. 58-61.
8.Ковалева Т.А., Башарина О.В., Селеменев В.Ф. Исследование структуры глюкоамилазы методом ИК спектроскопии // Тезисы докладов VII Всесоюзной конференции по спектроскопии биополимеров. – Харьков, 1991. – С. 134-135.
9.Ковалева Т.А. Кинетико-термодинамические аспекты катализа полисахаридов свободными и иммобилизованными амилазами // Биофизика. - Т.45, N3, - 2000 - C. 439-444.
10.Aleshin A., Firsov L., Harris E.,
Honzatko R. Refined structure fro the comlex of 1-deoxynojirimycin with
glucoamylase from Aspergillus awamori Var. X100 to 2.4 angstrom resolution
// Biochemistry. - V. 32. – 1993. –P.1618-1825.